アミノ酸配列解析 の履歴(No.2)
- 履歴一覧
- 差分 を表示
- 現在との差分 を表示
- ソース を表示
- アミノ酸配列解析 へ行く。
- 1 (2012-03-16 (金) 04:02:09)
- 2 (2012-03-16 (金) 09:28:02)
2-4連アミノ酸のSOM解析によるタンパク質の機能推定†
塩基配列解読技術の発達に伴い、ゲノム塩基配列の解読は飛躍的に加速しており、ヒトを始めとする真核生物から大腸菌や乳酸菌などの原核生物まで750種を超える生物種のゲノム配列解読が完了し、公開されており、3,000近くのゲノムプロジェクトが全世界で進行中である。塩基やアミノ酸配列の相同性検索法は、遺伝子やタンパク質の進化や系統の解析だけでなく、ゲノムが解読された際の各タンパク質の機能推定に不可欠の技術として普及し、バイオインフォマティクスの基本手法となった。この有用性が明らかになる一方で、新規性の高いゲノムが解読された際には、配列相同性検索でタンパク質の機能が推定できない遺伝子は半数近くに及ぶことも明らかになった。
タンパク質の機能については、機能部品類の3次元上での立体配置が重要であり、同一ないしは類似の機能を持つタンパク質間でも、アミノ酸の1次元配列上での全域に渡る相同性を見付けられない例が多い。この視点から、X線結晶構造解析やNMR法でタンパク質の3次元構造を決定し、機能既知タンパク質との高次構造上の類似性で機能を推定する大規模なプロジュクトが推進されている。しかしながら、費用や労力ならびに技術上の限界から、今後ますます急増する膨大な数の機能未知なタンパク質類の機能推定には不十分と考えられる。配列相同性検索を補完する、異なった原理に基づくタンパク質の機能推定法の確立が急務と言える。
我々は、これまでゲノムに潜む種固有の特徴を解明することを目的に、大量かつ多次元データの2次元や3次元での視覚化とクラスタリング法として、ヘルシンキ大学のコホネン博士[1]により開発された、教師なし学習アルゴリズムである自己組織化マップ法(Self-Organizing Map, SOM)の長所を生かしながら、再現性のある分類結果を得るアルゴリズムに変更し、「一括学習型自己組織化マップ法(BLSOM)」として提案し、ゲノム配列解析に適用してきた。
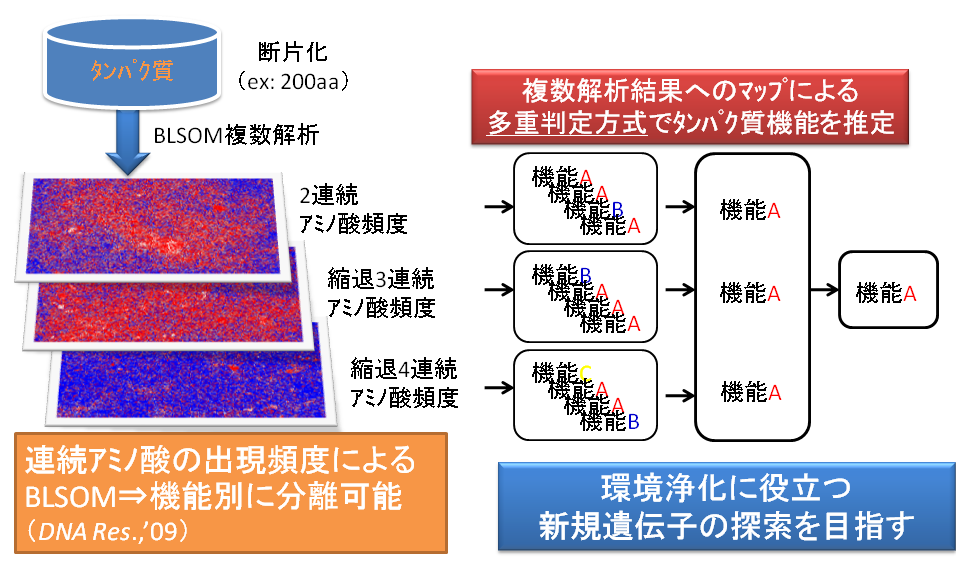
本研究では、BLSOM法をタンパク質に適用し、タンパク質の構造や機能モチーフを構成する部品である構成部品(オリゴペプチド)の使用頻度の類似度に基づくクラスタリングを基礎にしたアライメントフリーな機能推定法を確立した(図1)。
図1 連続アミノ酸組成に基づくBLSOMを用いたタンパク質機能推定法の概要図
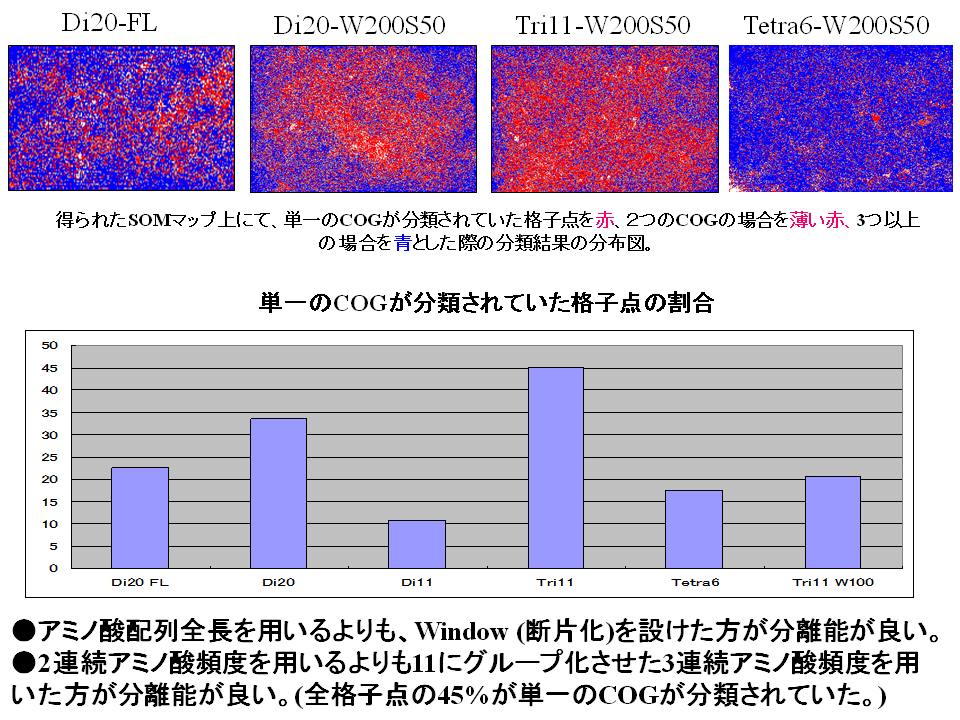
我々はタンパク質の2連や3連アミノ酸頻度を対象にしたBL-SOM解析を実施した。本解析では原核生物を中心とした機能カテゴリデータベースであるCOG (Cluster of Orthologous Gruop)を対象にして、機能が既知な機能カテゴリー(COGID数は2,853:配列数は113,738)を解析に用いた。2連アミノ酸頻度、20のアミノ酸を物理化学的な類似性で11のカテゴリーに集約した上での3連アミノ酸頻度、6のカテゴリーに集約した上での4連アミノ酸頻度に着目して、各タンパク質のアミノ酸配列の全長と、200アミノ酸のwindowを設けて50アミノ酸のstepで移動させた場合を試みた。Windowを設けることで、通常の大きさのタンパク質と大型タンパク質とを同時に解析することが可能となる。結果を図2に示す。図2では、アミノ酸配列全長を用いた2連続アミノ酸組成解析 (Dipep-FL (Full-Length))、断片化サイズ200aa (step 50aa)での2連続アミノ酸組成解析 (Dipep-W200S50)、ならびに断片化サイズ200aa (step 50aa)での集約11アミノ酸の3連続アミノ酸組成解析 (Tri11-W200S50)、集約6アミノ酸の4連続アミノ酸頻度解析(Tetra6-W200S50)でのBLSOMマップを示した。
図2 様々な解析条件におけるBL-SOMマップの分布図
同一のCOGに属する配列のみが分類されている点を赤とし、2種類のCOGが混在していた場合を薄い赤、3つ以上のCOGが混在していた場合には青とした。図2より、アミノ酸配列全長を用いるよりも、Window (断片化)を設けた方が、分離能が良く、2連続アミノ酸頻度を用いるよりも11に集約させた3連続アミノ酸頻度を用いた方が、分離能が良いことが判明した。また、断片化サイズ200aa (step 50aa)での集約11アミノ酸の3連続アミノ酸組成解析結果について、12の機能カテゴリの例を抜き出し、SOMマップ上での分布を見た結果を図3に示した。機能カテゴリごとに高い頻度で分類されていることが明らかである。
図3. 機能カテゴリごとのBLSOMマップ上での分布図
機能カテゴリごとに高い頻度で分類されていた結果を基に、機能が未知なタンパク質の機能推定を行った。近年、難培養性微生物類のゲノムDNA混合物を環境試料から培養過程を経ずに直接抽出し、混合ゲノムを対象とした配列決定法(メタゲノム解析)が普及しつつある。このめたメタゲノム解析由来の配列は新規性が非常に高く、遺伝子領域に関するアノテーションもほとんどついておらず、利用価値が低いままに国際配列データベースに登録されている。
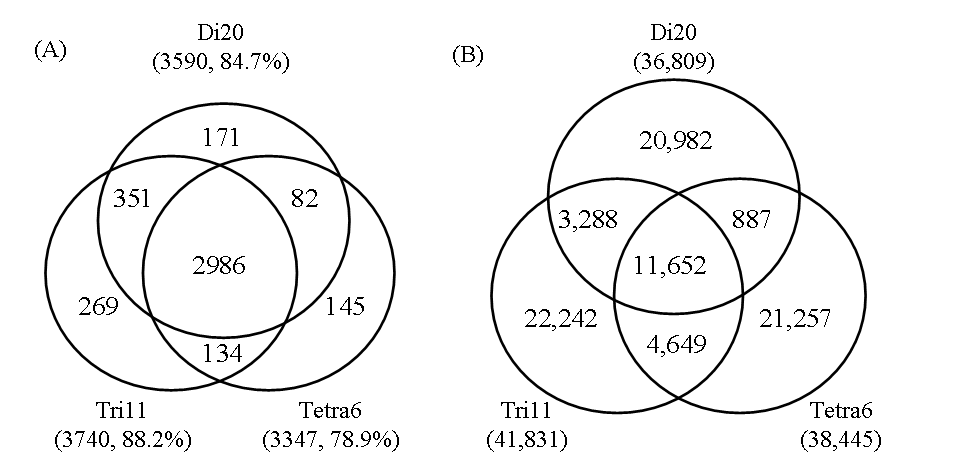
我々は以前に、メタゲノム解析由来のゲノム断片の系統推定法を確立し、報告している。今回開発した手法を用いて、メタゲノム解析由来のタンパク質配列を対象に、機能推定を実施し、本手法の検証を実施した。検証用のタンパク質データとして、Venterらによって報告されたSargasso海由来のメタゲノム配列中のタンパク質配列で既知遺伝子と高い相同性が得られたタンパク質配列(4240配列)を用いた。これらのタンパク質配列を用いて、断片化サイズ200aa (step 50aa)での2連続アミノ酸組成解析 (Dipep-W200S50)、ならびに断片化サイズ200aa (step 50aa)での集約11アミノ酸の3連続アミノ酸組成解析 (Tri11-W200S50)、集約6アミノ酸組成解析(Tetra6-W200S50)によって得られたBLSOMマップへ分類を行い、BLSOMマップ上に分類されていたアミノ酸配列のCOGの機能カテゴリを基に機能推定を実施し、 相同性で推定されたCOGの機能カテゴリとBLSOMマップ上への分類によって推定されたCOGの機能カテゴリが一致するかどうかを比較した結果を図4に示す。
図4相同性で推定されたCOGとSOM解析により推定されたCOGとの比較結果
その結果、Tri11-W200S50では、比較に用いた配列の85%近くが相同性検索にて推定されたCOGの機能カテゴリと一致しており、3つの解析条件で共通に一致していた割合も70%と高い割合で一致していた。機能未知なタンパク質を対象により確かな機能推定を行うためには、単一のBLSOM解析条件下での推定結果のみを用いるのではなく、複数の推定結果をもとに機能推定を実施する方が良いと考えられる。また、同様の解析を相同性検索でCOGへの分類が困難な93,598の200aa以上のタンパク質配列に対し行い、その30%近くがBLSOMによってCOGと関係付けられる可能性が高い。
このように、オリゴペプチド組成に基づくBLSOM解析によって、タンパク質の機能を反映した分類が可能であり、類似した機能を持つタンパク質の関係も見ることができ、機能未知タンパク質に対し、相同性検索とは異なった視点(アラインメントフリー)でのタンパク質機能推定が可能である。
詳しくは、Abe et al. DNA Res., 2009をご覧下さい。†
配列相同性検索との比較から得られる遺伝子構造の分子進化学的解析†
上述のタンパク質の機能推定は、タンパク質を構成する部品に着目した方法であり、モチーフサーチと似た原理に基づいている。指摘すべき点は、機能モチーフ以外にも、タンパク質の高次構造形成に必要な要素部品の配列類の構成をも加味している。タンパク質の機能発現に重要な機能部位については、3次元上での部位間での相互配置が重要であり、1次元配列の相同性検索では有意な相同性を見付けられない可能性がある。その意味でも、要素部品(オリゴペプチド)の出現頻度の類似度を基礎にして、機能未知のタンパク質と機能既知のタンパク質とを関連付ける方法論の開発は興味深い。勿論、この技術を確立するには、克服すべき課題も多い。しかしながら、相同性検索のみでは、ゲノム解読から得られる遺伝子候補群について、半数にも及ぶ例でタンパク機能が未知残されている現状を考えると、配列相同性検索に依存しない方法論の導入の意義は大きい。本申請の研究の目的は、配列相同性検索法を補完する新技術の確立にある。機能が未知に残されてきたタンパク質類について、機能が既知のタンパク質とSOM法で有意な関連付けが可能になった場合には、それらについての配列相同性検索を行い、構成部品に着目したSOM法と相同性検索で差異の生じた原因を知る。単純な配列相同性検索に、タンパク質の高次構造の類似度を加味したことで、系統や機能推定の信頼度が上昇したのと同様に、要素部品(2~4連アミノ酸)の構成の類似度を加味することで、系統や機能推定の信頼度の上昇や適応範囲の拡大が期待できる。個々の遺伝子の分子進化過程に有用な知見が得られる可能性も考えられる。